ActiveNews se confruntă cu cenzura pe rețele sociale și pe internet. Intrați direct pe site pentru a ne citi și abonați-vă la buletinul nostru gratuit. Dacă doriți să ne sprijiniți, orice DONAȚIE este binevenită. Doamne, ajută!
Știm că unul din obiectivele
celei de-a patra revoluții industriale este editarea genică.
https://www.weforum.org/about/the-fourth-industrial-revolution-by-klaus schwab,https://www.weforum.org/agenda/2019/06/where-will-evolution-take-us-in-the-fourth-industrial-revolution/,https://www.weforum.org/agenda/2023/02/crispr-gene-editing-better-world/.
Organizația Mondiala a Sănătății a publicat în anul 2021 câteva broșuri pe tema
editării genice https://www.who.int/teams/health-ethics-governance/emerging-technologies/human-genome-editing.
Observăm că se vorbește din ce în ce mai mult despre augmentarea umană https://www.gov.uk/government/publications/human-augmentation-the-dawn-of-a-new-paradigm.
Este clar că se întâmplă ceva major în acest tetoriu al geneticii, mai ales
după ce am avut de-a face cu injectarea la nivel global cu produsele genice
anti-covid.
S-a afirmat cu convingere de
către „specialiști” că așa-numitele vaccinuri ARNm anti-covid nu se pot integra
în ADN. Recent am găsit un articol care demonstrează contrariul și care descrie
chiar mai multe mecanisme prin care se poate produce acest fenomen. In plus, în
acest articol sunt descrise și modalitățile prin care proteina spike poate
determina alterarea ADN-ului, favorizând apariția cancerelor. Am tradus acest
articol dificil , cu multe noțiuni de genetică, unele fiind noi și pentru mine.
Am considerat că este foarte important să cunoaștem aceste opinii solid
argumentate științific pentru că se dorește ca toate vaccinurile și multe din
terapiile viitorului să folosească tehnologia ARNm. Acest articol poate fi găsit în engleză, ca
rezumat la link-ul https://europepmc.org/article/PPR/PPR539081și full-text la link-ul https://d197for5662m48.cloudfront.net/documents/publicationstatus/89121/preprint_pdf/7a197d8835afa569325ddec4d62f61f8.pdf.
Mecanisme potențiale de integrare în genomul uman al
codului genetic al ARNm-ului vaccinului anti SARS-CoV-2
Anthony M
Kyriakopoulos(1), Peter A Mccullough (2), Greg Nigh(3)și Stephanie Seneff(4)
1 Director și
șef al departamentului de cercetare și dezvoltare, Departamenul de Cercetare și
Dezvoltare, Laboratorul de biotehnologie Nasco AD
2 Consilier
medical principal, Fundația Adevărul pentru sănătate
3 Immersion
Health, Portland, OR 97214, SUA.
4 Cercetător
științific principal, Laboratorul de informatică și inteligență artificială, Institutul
de Tehnologie din Massachusetts
1 septembrie
2022
Rezumat
Context
Descoperirea unei secvențe încorporate în ADN uman care era
aproape identică cu o secvență a genomului SARS-CoV-2 și identificarea unei
integrări plauzibile a ARN-ului SARS-CoV-2 în ADN-ul uman prin activitatea reverstranscriptazei
endogene exprimată de Elementul Nuclear Lung Intercalat (LINE)-1 (17% din ADN
uman) au generat îngrijorări cu privire la siguranța pe termen lung a
vaccinării ARN mesager (ARNm). Date recente demonstrează că secvențele de ARN
ale SARS-CoV-2 pot fi transcrise în ADN și pot fi integrate în mod activ în genomul
celulelor umane afectate, prin intermediul retrotranspozonilor.
În plus, la unii pacienți infectați cu SARS-CoV-2, există
dovezi de integrare a unei secvențe mari a SARS-CoV-2 și generarea ulterioară
de transcripte chimerice umane de SARS-CoV-2.
Rezultate
În acest articol, se reconsiderăă rolul potențial al
elementelor genetice mobile în etiopatogenia bolilor cardiovasculare,
neurologice, imunologice și oncologice și posibilitățile de afectare a ADN-ului
uman prin vaccinarea SARS-CoV-2. Celulele stem umane vulnerabile, precum și
gametocitele pot fi, probabil, primele ținte pentru acțiunea nedorită a
ARN-ului. Având în vedere numeroasele manipulări genetice ale ARN care codifică
glicoproteina spike a SARS-CoV-2 din vaccinuri, manipulările concepute pentru a
crește stabilitatea și eficiența traducerii proteinei spike – rămân multe
incertitudini cu privire la posibilele perturbări ale fiziologiei și
homeostaziei celulare. Consecințele prezise reprezintă riscuri grave pentru
sănătatea umană, care necesită clarificări.
Concluzie
Este nevoie urgentă de evaluări suplimentare ale toxicității
pentru a cuantifica potențiala interferență cu procesele canonice ale ADN-ului
care ar putea avea un impact negativ asupra populației vaccinate cu ARNm.
1. Introducere
Un argument major în favoarea siguranței pe termen lung a
vaccinării cu COVID-19, așa cum a fost analizat de Pardi et al. în 2018 [1], a
fost tratat de autori după cum urmează: ”La persoanele vaccinate, riscurile
teoretice de infecție sau de integrare a vectorului în ADN-ul celulei gazdă nu
reprezintă o preocupare pentru ARNm. Din motivele de mai sus, vaccinurile cu
ARNm au fost considerate ca fiind relativ sigure”. Cu toate acestea, se pare
că nu este așa, siguranța neputând fi prezumată. În ADN-ul uman se află
încorporată o secvență identică în proporție de 94,6 % (117 pb) a SARS-CoV-2,
care este localizată în cromozomul 1p, în regiunea intronică a genei netrin G1
(NTNG1), după cum au demonstrat Lehrer și Rheinstein [2]. Această secvență a
fost descoperită în timpul fazei incipiente a pandemiei COVID-19 și corespunde
unei secvențe orf1β aproape identice cu a genei SARS-CoV-2, care este similară cu secvența
glicoproteinei spike (principala cauză a patologiei COVID-19) [2-4]. Mai exact, secvența omologă umană
SARS-CoV-2 se potrivește cu o secvență orf1β a proteinei nestructurale (nsp) 14
(care este o exonuclează) și a nsp 15 (care este o endoribonuclează) a
virusului [4].
Deși SARS-CoV-2 este un virus ARN monocatenar și nu un
retrovirus, integrarea sa în genomul uman se poate realiza în diferite moduri,
fie a) prin intermediul reverstranscriptazei inverse endogene Long Interspersed
Nuclear Elements-1 (LINE-1) [5,6] sau b) prin intermediul transcriptazei inverse
umană recent recunoscută, polimeraza theta, a cărei activități de
revers-transcripție este comparabilă cu cea a retrovirusului imunodeficienței
umane (HIV) [7], sau c) prin intermediul mecanismelor de reparare ale ADN-ului
defect dublu catenar [8,9]. Copiile ADN ale virusului sunt capabile să
se integreze în mai multe situsuri ale cromozomilor umani, așa cum este descris
în articolele lui Katsourakis și Glifford 2010 [10] și Geuking et al. 2009
[11], deși ARN-ul SARS-CoV-2 nu ar fi trebuit să fie revers-transcris în
celulele umane.
Copiile secvenței SARS-CoV-2 cel mai frecvent
integrate în ADN uman sunt cele apropiate
de regiunile netranslatate (UTR) 5′ și 3′, arătând o preferință pentru
secvențele vecine cu promotorii și cu capetele poli(A) [4]. Secvența
SARS-CoV-2 LINE-1 RT de integrare în ADN uman pare să nu fie aleatorie, ci țintește
mai degrabă situs-urile umane asociate exonilor [5]. Integrarea corespunde
nucleocapsidei subgenomice de dimensiuni complete (non spike) a secvențelor
SARS-CoV-2, care poate fi exprimată în transcripte chimerice (virus-gazdă) în
celulele umane [5].
Secvențele nucleocapsidelor și ale proteinei spike sunt cele
mai bogate domenii genomice ale SARSCoV-2 în ceea ce privește substituțiile
nesinonime și, prin urmare, sunt de mare interes medical și epidemiologic [12].
În această privință, o reactivitate robustă a celulelor T (limfocitelor CD4+ și
CD8+) împotriva nucleocapsidei și a proteinei spike a fost înregistrată până la
60 % din probele provenite de la o populație neexpusă la SARS-CoV-2 în timpul
debutului pandemiei COVID-19 [13,14]. Această memorie imunitară naturală
importantă împotriva SARS-CoV-2 a fost atribuită fie infecțiilor anterioare cu coronavirusul
răcelii comune [13,14], fie transmiterii anterioare a coronavirusurilor între
specii, de la animale la oameni [15,16].
Integrarea întregului genom sau a unor secvențe
genomice ale altor retrovirusuri sau virusuri cu ARN sau ADN în genomul uman
este dovedită și acestea se pot fixa în cromozomi după mai multe generații[10]. Ca atare, prezența de ARNm sintetic [1] în vaccinurile ARNm, având
secvențe din proteina patogenă spike a SARS-CoV-2 în imediata apropiere a cozii
poli(A), înseamnă, de asemenea, că acesta are toate premisele pentru a se
insera în ADN-ul uman. În plus, o atenție deosebită în procesul de
fabricare a ARNm sintetic a fost acordată modificărilor nenaturale, cum
ar fi conversia tuturor uridinelor în metilpseudouridine, cu scopul de a
proteja ARNm de degradare [1]. Această longevitate sporită în interiorul celulei
crește probabilitatea de transcriere inversă și de încorporare în ADN prin
diverse mecanisme care implică elemente mobile [17]. Prin urmare, sunt
revizuite posibilele mecanisme de interferență genetică umană și consecințele
asupra sănătății umane. În mod remarcabil, s-a demonstrat în experimente
pe șoareci că sperma mamiferelor este capabilă să translateze ARN-ul mesager
exogen în ADN, să grupeze ADN-ul în plasmide și să elibereze aceste plasmide în
mediul local în timpul fertilizării. Un ovul fertilizat poate prelua plasmidele,
le poate păstra pe tot parcursul vieții fetale precum și după naștere, pe tot
parcursul vieții. Ele pot fi chiar transmise mai departe
generațiilor viitoare. Aceste plasmide pot rămâne autonome și
sunt capabile să își cloneze ADN independent de genomul uman [18]. Prin urmare,
este posibil ca un astfel de proces să aibă loc în urma vaccinării cu ARNm,
ceea ce ar determina sintetiza proteinei spike de către celulele copilului, al
cărui sistem imunitar sistem imunitar ar considera proteina spike ca fiind o
autoproteină, fapt cu consecințe necunoscute.
2. Transcripția inversă mediată de LINE-1 a ARNm al
vaccinului
Cercetătorii din Suedia au efectuat un studiu in vitro pe o
linie celulară de carcinom hepatic uman (celulele Huh7) expusă la vaccinul
Pfizer BioNtech BNT162b2, examinând dacă aceste celule au capacitatea de a
transforma ARNm din vaccin în ADN [19]. Autorii au constatat că celulele au
preluat ușor și spontan nanoparticulele de ARNm și au răspuns la expunere prin
up-reglarea LINE-1. Un test imunohistochimic a arătat că nivelurile de
LINE-1 au crescut în nucleu ca răspuns la nanoparticulele de ARNm. În
mod alarmant, aceștia au verificat că o regiune ARNm reporter de 444 perechi de
baze (amplicon) a fost ușor revers-transcrisă intracelular în ADN după 6 ore de
la expunere.
Studiile au arătat că ARNm din vaccinuri intră în sistemul
limfatic și este în cele din urmă preluat de celulele din mai multe organe, ficatul
fiind al doilea după splină în ceea ce privește concentrația detectată[20]. Aldén et al. au sugerat că celulele hepatice ar putea expune
proteina spike pe suprafața lor și, în acest fel, ar putea induce un atac
autoimun asupra celulelor prin anticorpi [19]. Aceasta ar explica
cazurile de hepatită autoimună apărute după vaccin [21-23].
ARNm din vaccinuri a fost modificat pentru a avea o coadă
lungă de poli(A), care ajută la atât la facilitarea traducerii în proteine, cât
și la creșterea timpului de supraviețuire a ARNm.
Cu toate acestea, prezența unui număr mare de molecule de
ARNm cu cozi lungi de poli(A) crește probabil expresia proteinei de legare a
poli(A) (PABP), pentru a răspunde nevoilor acestor molecule de ARNm. S-a
constatat că PABP este esențială pentru retrotranspoziția eficientă a LINE-1,
iar reducerea PABP scade foarte mult activitatea LINE-1 [24].
Proliferarea LINE-1 implică un ciclu de viață complex care
începe cu transcripția ARNm al ARN polimerazei II (Pol II). ARNm este tradus în
cele două ORF-uri ale sale în citoplasmă.
ORF-urile formează o particulă de ribonucleoproteină (RNP)
care se transferă apoi în nucleu pentru traducerea ARN-ului în ADN și
integrarea în genom. S-a emis ipoteza că PABP acționează ca o proteină de
escortă care poate transporta RNP către nucleu [25]. Prin urmare, mecanismul
prin care ARNm din vaccinuri crește activitatea LINE-1 ar putea fi prin
creșterea PABP.
3. Cancerul crește riscul de retrotranscriere a ARNm al
Spike?
Modificarea epigenetică care implică metilarea citozinei în
insulele CpG este un factor important în reglarea expresiei genice. Se
estimează că mai mult de 90% din toate 5-metilcitozinele se află în insulele
CpG ale transpozonilor, adică în elementele nucleotidice intercalate lungi și
scurte (LINE și SINE). De fapt, gradul de metilare a LINE-1 este considerat un
marker surogat al metilării globale a ADN-ului. Hipometilarea promotorului
LINE-1 activează expresia acestuia. Nivelurile ridicate ale activității
LINE-1 sunt asociate cu multe tumori, inclusiv cancer de sân, esofagian, de
colon și carcinomul pulmonar cu celule scuamoase. LINE-1 poate media inactivarea genelor
supresoare tumorale și promovează proliferarea celulară și invazia[26]. Experimentul realizat de Aldén et al. care demonstrează transcrierea
inversă a ARNm al proteinei spike a fost realizat pe celule de carcinom hepatic
uman crescute în cultură. Cancerul hepatic reprezintă 9% din din toate tipurile
de cancer la nivel mondial, iar 80% din cazuri sunt diagnosticate ca fiind
carcinom hepatocelular. În mod intrigant, a fost găsită o legătură între
retrotranspozonii LINE-1 și hepatita B sau infecția cu hepatita C. Câteva
transcripții chimerice LINE-1 cu gene gazdă sau virale sunt găsite în
carcinomul hepatic cauzat de virusul hepatitei. În plus, s-a demonstrat că
retrotranspoziția LINE-1 endogenă activează căi oncogene [27]. Aceste
observații sugerează că vaccinurile cu ARNm ar putea induce sau accelera
avansarea carcinomului hepatocelular (HCC) la oamenii expuși printr-un proces
similar, prin creșterea activității LINE-1. În acest sens, dezvoltarea
HCC este legată de infecția cronică cu virusul hepatitei C (VHC – un virus ARN
cu catenă pozitivă) [27]. Prin intermediul mecanismelor alternative de
retrotranspoziție LINE-1 [28], pare posibil ca ARN-ul VHC să fie transcris
invers. În mod complementar, producția de clone de ADNc (ADN complementar) din ARN-ul VHC a fost obținută
la modele animale primate [29], iar secvențe ale genomului VHC sunt ușor de
detectat la alte specii de mamifere [28].
Mai mult, când gradul de hipometilare a ADN-ului LINE-1 din
celulele tumorale hepatice a fost comparat cu cel al celulelor normale
adiacente, rezultatele (48,6 % față de 71,7 % metilare) au fost foarte
semnificative (p < 0,0001) [30]. Un studiu privind cancerul de colon a
arătat că LINE-1 a fost hipometilat chiar și în celulele țesutului normal
adiacent tumorii, această constare fiind asociată cu rezultate mai proaste în
rândul pacienților cu cancer [31]. LINE-1 hipometilat și puternic
exprimat a fost, de asemenea, găsit în boli autoimune, cum ar fi lupusul
eritematos sistemic, sindromul Sjögren și psoriazisul [26]. Având
în vedere că expunerea la ARN-ul SARS-CoV-2 a provocat o expresie crescută a
LINE-1 la pacienții infectați [5], această sugerează, de asemenea, că vaccinul
genetic ARNm ar putea determina un risc crescut de apariție a cancerului sau a
unei boli autoimune prin intermediul unei posibile integrări în ADN mediate de
LINE-1. Acest lucru poate fi, de asemenea, de așteptat să accelereze progresia
bolilor mai sus menționate.
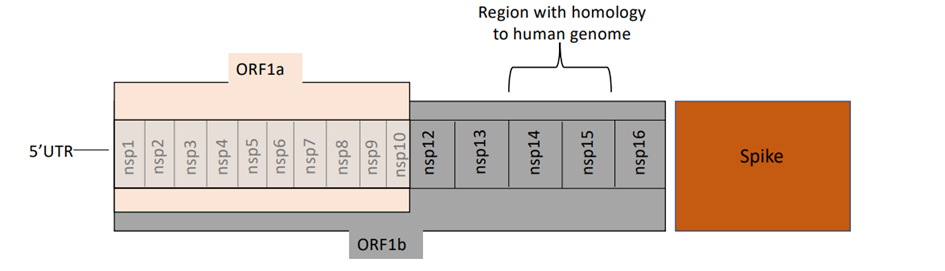
Figura 1: Ilustrare a segmentului din genomul SARS-CoV-2
care este aproape identic cu o secvență genetică umană. Adaptat din figura 2,
Rastogi et al., 2020 [32].
Elementele genetice mobile și etiopatogenia bolilor
neurologice
Segmentul din SARS-CoV-2 care este aproape identic cu o
secvență genetică umană se află în nsp14 și nsp15 din genomul viral, doar nsp16
(o secvență de 904 pb) separându-l de secvența proteinei spike, așa cum este
schematizat în figura 1. [1,4]. Acest segment este încorporat în orf1β al
SARS-CoV-2. Alte ORF-uri virale, codificate ca elemente endogene, sunt
exprimate ca ARNm în celulele umane [10]. Secvența genomică umană care este omologă cu genomul SARS-CoV-2 este
localizată în gena NTNG1 [2, 4].
Este important faptul că tulburările dezvoltării neuronale
asociate cu anomalii genetice în cadrul
genelor NTNG1 și NTNG2 se dovedesc a fi responsabile pentru fiziopatologia
schizofreniei [33,34].
O multitudine de proteine de legare a ARN sunt implicate în
controlul transcripției [35].
Chiar dacă doar o mică parte din ARN sintetic ajunge în
celule, prezența ARNm sintetic în vaccinuri, chiar și la concentrații de 30 ug
și 100 ug (un minim de 40 de trilioane de molecule de ARNm sintetic) poate
produce complexe de legare ARN-proteine care controlează transcripția. De
exemplu, acest lucru este important atunci când proteina de legare poate fi
topoizomeraza 3β (cu proprietăți biologice de control al funcțiilor mentale, de
îmbătrânire și de neurodezvoltare), deoarece această enzimă specifică formează
un complex foarte conservat și important din punct de vedere medical cu încă o
proteină, Tudor-domain containing 3 (TDRD3) [36]. Acest complex puternic poate
interacționa cu histonele, ARN monocatenar, ADN, factori de translație și
polimeraza II. Acest lucru poate provoca defecte nefiziologice de
neurodezvoltare și îmbătrânire la om [36].
În timpul studiilor de descoperire a relațiilor proteină SARS-CoV-2
– proteină umană, au fost evidențiate 332 de interacțiuni de încredere ridicată
între cele două specii [1]. Aceste interacțiuni au demonstrat de fapt
eficacitatea promițătoare a clorochinei și a unui medicament antipsihotic,
haloperidol, împotriva SARS-CoV-2. Cu toate acestea, aceste numeroase
interacțiuni proteină-proteină complică și mai mult posibila expresie a proteică
a secvențelor SARS-CoV-2 din ADN uman și interacțiunile acestora prin
intermediul Retroviralelor Endogene Umane, al revers-transcriptazelor Alu și
LINE-1 codificate de ADN și al altor proteine endogene umane [37]. S-a
demonstrat că astfel de interacțiuni au consecințe grave în bolile neurologice
[38]. Acest lucru poate fi și mai important pentru pacienții deja infectați cu
SARS-CoV-2, care primesc apoi secvențele proteice spike în vaccinurile ARNm și
care au deja secvențe SARS-CoV-2 reverstranscrise, răspândite în organele
sensibile, cum ar fi sistemul nervos central [1,4,5]. Acest lucru poate avea
consecințe foarte importante pentru acei pacienți care suferă, de asemenea, de
boli neurodegenerative preexistente. [38]. Deja, există rapoarte
emergente privind asocierea vaccinării ARNm COVID-19 cu accelerarea a bolii
Parkinson [39,40] și a bolii prionice [41].
Cercetările recente arată prezența persistentă în sânge până
la 15 luni post infecție a subunității S1 (S1) a spike-ului SARS-CoV-2 care
este capabilă să traverseze bariera hemato-encefalică, probabil prin exozomi,
la pacienții care suferă de sechele post-acute ale infecției cu SARS-CoV-2
[42]. Cu toate acestea, această constatare necesită investigații suplimentare
pentru a se stabili dacă proteina S1 este purtată în mod persistent de către
monocitele non-clasice CD14lo, CD16+ pentru o perioadă lungă de timp perioadă
lungă de timp sau dacă, în schimb, prezența S1 este rezultatul producției ADN-ului
endogen, deoarece posibilitatea persistenței virusului întreg în celule a fost
exclusă în acest studiu [42].
Retrotranspoziția poate explica, de asemenea, prezența
durabilă atât a ARNm, cât și a proteinei spike în centrii germinativi ai
ganglionilor limfatici până la 60 de zile după vaccinare [43].
4. Vaccinarea cu ARNm SARS-CoV-2 poate provoca
interferențe cu genomul uman
ADN uman ca și alte ARN-uri virale
Moleculele de ARN au capacitatea de a-și modifica în
mod spontan secvențele și, chiar și atunci când sunt fragmentate, să dirijeze
sinteza copiilor lor [44]. Recombinarea ARN-ului [45] și transmisibilitatea
prin intermediul spermatozoizilor [18] sau prin reacții de metateză la
următoarea generație de celule reprezintă unul dintre obstacolele majore care
trebuie depășite în aplicarea tehnologiei ARNm pentru vaccinarea împotriva
bolilor infecțioase [1]. Repetițiile terminale lungi (LTR) din cadrul Retrovirusurilor
Umane Endogene (HERV) [37,46] conțin secvențele necesare de promotori, enhanceri
și cozi poli(A) care să reverstranscrie o secvență de ARN străin într-un dsADN
și, ulterior, ca în cazul ARN-ului SARS-CoV-2, să integreze multiple fragmente
în diverși cromozomi umani [1,4,7].
Prin urmare, LTR-urile dispun de toate mecanismele, alături
de enzimele necesare pentru reverstranscriere [7] și integrare în ADN-ul uman,
pentru a codifica învelișul viral, nucleocapsida și capsida matricei [38] și
pentru a produce particule recombinate virale noi având secvențe chimerice
(gazdă și virală). Secvențe chimerice similare au fost detectate în liniile
celulare infectate cu SARS-CoV-2 [5]. Deoarece retrotranspozonii LINE-1 sunt
amplificați în timpul vieții embrionare timpurii [47], acest lucru constituie o
probabilitate ca celulele dendritice circulante derivate din celulele stem
hematopoietice și celulele reproductive (ovocite și spermatozoizi) să fie
afectate. Acest lucru este valabil chiar și în cazul unor doze mici de ARNm
în vaccinuri, în care secvențele de ARN ale proteinelor spike de SARS-CoV-2 cu
durată lungă de viață ar putea fi revers-transcrise la intrarea în celulă și,
ulterior, ar putea fi codificate în linii germinale. Aceasta poate
determina o producție suplimentară de secvențe de proteine spike, dincolo de
cele prevăzute inițial prin expresia endogenă [1]. În acest sens, asamblarea de
virioni purtători de secvențe chimerice SARS-CoV-2 este o consecință probabilă
pe termen lung [5,7]. Inserțiile funcționale în cadrul secvențelor HERV, altele
decât cele determinate de evoluție [10], pot activa genele HERV și LTR, silențioase
epigenetic . Cea mai mare îngrijorare este că acestea pot deveni active
și pot juca un rol cauzal în autoimunitate, tumorigeneză și în progresia altor
boli [48].
Prin acest tip de interferență foarte plauzibilă a ADN-ului,
prin secvențele ARNm ale SARS-CoV-2 [1,4,5], pot fi activate și regiuni ale
ADN precum cele ale elementelor HERV-K, numite regiuni HERV-K ale virusului
tumorii mamare la om și șoarece (HML-2) [46,48]. Un astfel de fenomen a
fost deja dovedit în cazul altor virusuri ARN non-retrovirale [10], precum și a
altor secvențe de coronavirus [4]. Regiunea HERV-K (HML-2) conține
singură mai mult de 90 de segmente de provirus împrăștiate în întreg ADN-ul transcripțional
activ și acestea pot fi carcinogenice, determinând melanoame si
teratocarcinoame [49].
Aceste evenimente genetice nefericite se pot întâmpla prin
simpla perturbare a mecanismelor naturale anticancerigene de apărare ale
gazdei, dezvoltate de-a lungul a milioane de ani prin co-evoluția gazdei cu schimbul
de material genetic viral și al diseminării în întreg genomul uman ca o linie
de apărare a sănătății [49].
5. Potențialul de inducere a oncogenezei și a
metastazelor: Rolul celulelor stem
Pentru a obține rezultate optime ale expresiei proteice în
timpul dezvoltării vaccinării intradermice cu ARNm sintetic, a fost necesară o
secvență de macromolecule de cel puțin 900 bp [50]. Greutatea moleculară medie
a ribonucleotidelor monofosfați este de 339,5 g/mol (MW) [51]. Acest lucru face
ca dozele de 30 µg și 100 µg de vaccinuri cu ARNm sintetic să pară, la prima
vedere, extrem de mici pentru a fi capabile de interferență genetică cu
celulele umane [50]. În cazul celulelor care nu se divid, se pare că riscul de
mutageneză inserțională este scăzut [50].
Cu toate acestea, ARNm sintetic, chiar și în câteva minute
după vaccinare, diseminează rapid de la locul injectării în rețeaua de drenaj a
ganglionilor limfatici învecinați [52]. Nișele ganglionilor limfatici răspândite
larg în întregul organism conțin celule stem nediferențiate precursoare care
primesc semnale de proliferare în condiții de stres și, prin urmare, diviziunea
mitotică a acestor celule este ridicată [53].
Celulele stem umane sănătoase (CSH) au o capacitate importantă
de diviziune mitotică accelerată care le conferă o capacitate sporită de transformare
în celule stem canceroase în comparație cu celulele stem de la alte specii. De
fapt, datorită capacităților lor unice de a se regenera și de a forma țesuturi
ex vivo, întreaga tehnologie de editare a ARN a fost construită pentru a servi unor
scopuri curative [54]. De notat, editarea ARN a CSH realizată în laborator este
transmisă în mod robust și cu o frecvență ridicată de la CSH parentale la
următoarele generații de celule, care devin apoi celule stem modificate canceros.
Studiile de
laborator sugerează cu tărie că moleculele sintetice de ARNm conținute în
vaccinurile actuale cu ARNm ar putea induce modificări patologice în
mecanismele celulare stabilite pentru modificările pre-ARNm care au loc în
nucleul celulelor HSC din sistemul limfatic [53].
Editarea ARN-ului (modificări epigenetice și reglări
post-transcripționale) este un proces extrem de sensibil, ale cărui erori poate
determina malignitatea celulelor stem. Celulele stem au o orchestrare foarte
vulnerabilă a evenimentelor genetice ca răspuns la factori atât intrinseci (în
interiorul celulei), cât și extrinseci (în afara celulei) [54]. De asemenea,
s-a dovedit că apariția malignității din celule stem anterior sănătoase este
ușor de indus prin interferența microARN (miARN) endogen (reglaj epitranscriptomic)
în timpul editării ARNm [54]. În plus, deoarece stimularea activării,
diferențierii și proliferării este o activitate obișnuită a celulelor imune și ale
altor celule stromale localizate în ganglionii limfatici [53], riscul de
interferență a ADN-ului sau de perturbare epigenetică chiar și de către o
singură macromoleculă sintetică de ARNm care intră în celulă, nu poate fi
exclus.
6. Proteina Spike induce semnalul oncogenic prin
intermediul căii JAK/STAT3
Se poate demonstra cu ușurință că o celulă stem dintr-un
ganglion limfatic este vulnerabilă la oncogeneză prin influența glicoproteinei
spike, care este produsă de ARNm-ul din vaccin. Numeroase studii au
arătat că proteina spike este capabilă să inducă singură o supraexpresie a
citokinei proinflamatorii interleukina-6 (Il-6) [55-57].
Această citokină, la rândul său, induce fosforilarea
tirozinei STAT3, care apoi migrează în nucleu pentru a induce un răspuns
inflamator [55].
În nucleu, STAT3 se leagă de și activează promotorii unui
panel larg de gene care codifică proteine care induc proliferarea celulară, o
etapă esențială pentru tumorigeneză [58].
Hiperactivarea STAT3 apare în multe tipuri de cancer,
inclusiv în cazul leucemiei mieloide acute, a mielomului multiplu și a tumorilor
solide ale vezicii urinare, oaselor, sânului, creierului, colului uterin, colonului,
esofagului, capului și gâtului, rinichiului, ficatului, plămânului, ovarelor,
pancreasului, prostatei, stomacului și uterului [58].
Calea JAK/STAT3 promovează conversia celulelor stem
preleucemice umane în celule stem de leucemie mieloidă acută. Mecanismul
implică activarea enzimelor care deaminează adenozina din ARN-ul bicatenar,
transformând-o în inozină. Această clasă de enzime se numește adenozin
deaminază care acționează asupra enzimelor ARN (ADAR), acestea fiind sunt în
legătură strânsă cu cancerul [59]. Acestea induc o transformare a A în I în ARN-ul
dublu catenar care, în cele din urmă, are ca rezultat o codificare greșită a
adenozinei ca guanidină. Amorsarea Il-6 prin intermediul unui vaccin recent cu
ARNm ar putea accelera rata de mutație în ARNm al proteinei spike în timpul
unei infecții ulterioare cu SARS-CoV-2, în
mod direct, prin up-reglarea enzimelor ADAR. Acest lucru ar putea explica
apariția rapidă a variantelor rezistente ale SARS-CoV-2 în ultimele luni, în
urma inițiativei de vaccinare agresivă a populației [60].
Editarea A-la-I a ARN-ului bicatenar este un mecanism reglator
post-transcripțional care joacă un rol important în cancer. Un loc important în
care are loc editarea A-to-I este regiunea 3′-UTR a moleculelor de ARNm. Este
important faptul că o astfel de editare duce la o scurtare a segmentului
3′-UTR, ceea ce duce adesea la eliminarea situsurilor de legare pentru
miARN-urile care suprimă sinteza proteinelor. În acest sens, o astfel de
editare crește expresia proteică a genei afectate, ceea ce duce adesea la
carcinogeneză [61].
HSC au potențialul de a se diferenția și de a deveni orice
tip de celulă din organismul matur, iar interferența genetică insuficient
ghidată a celulelor stem poate duce la boli precum tulburările hematopoiezei,
precum și la cancer [62]. Celulele tumorale din circulația limfatică sunt
frecvent prezente la pacienții cu tumori maligne diagnosticate. Prezența
acestor celule poate fi constatată și la pacienții cu o afecțiune malignă
nediagnosticată. Aceste celule din circulația limfatică au un potențial
metastatic important din punct de vedere clinic în comparație cu celulele
maligne care circulă în sânge și care au scăpat din tumorile primare, iar acest
proces poate fi amplificat de orice interferență ARN. Deoarece ARNm sintetic
tinde să se disperseze și să se acumuleze în ganglionii limfatici regionali
[52], iar celulele maligne circulante în limfă au un mozaic de proliferare a
ciclului celulelor stem [48,54] și utilizează întregul sistem limfatic pentru a
se deplasa, atunci riscul potențial metastatic poate fi considerat ca fiind
ridicat în aceste cazuri [62].
7. Proteina Spike, inflamația, sincițiul, leziunile ADN
și senescența
Într-un experiment de laborator inteligent conceput, Meyer
și colab. au explorat ideea că expunerea epiteliului pulmonar la proteina spike
poate duce la eliberarea de mediatori care determină disfuncție endotelială
[63]. Acești cercetători au demonstrat că celulele epiteliale umane A549
transfectate cu spike au eliberat molecule inflamatorii care sunt
caracteristice unui fenotip secretor asociat senescenței (SASP), împreună cu o
creștere de 3 ori mai mare a speciilor reactive de oxigen (SRO). Mai mult,
nivelurile de SRO au crescut de aproximativ 2 ori în celulele endoteliale
expuse mediului de cultură prelevat de la celulele A549 producătoare de spike,
în comparație cu celulele de control transfectate cu plasmidă goală. Prin
urmare, celulele endoteliale răspund la semnalizarea din partea celulelor
epiteliale transfectate cu plasmidă de spike printr-un „răspuns de
senescență bystander” care poate duce la deteriorarea endotelială printr-un
proces paracrin. Senescența celulară a fost, de asemenea, asociată cu un nivel
crescut al moleculelor de adeziune endotelială care promovează legarea
leucocitelor la peretele vascular. O astfel de legare este un prim pas spre
extravazarea leucocitelor și invazia și inflamația ulterioară a țesuturilor.
Aceste rezultate sunt în concordanță cu cele ale unui alt
studiu care a investigat răspunsul celulelor epiteliale și endoteliale
bronșice cultivate la transfecția cu proteină spike, care a demonstrat, de asemenea,
că celulele au răspuns cu niveluri crescute de ROS, declanșând un răspuns
inflamator și în final, apoptoză [64].
O serie remarcabilă de lucrări realizate de o echipă mare de
cercetători din China a demonstrat în mod incontestabil că proteina spike
determină celulele care au receptori ACE2 (cum ar fi celulele
epiteliale pulmonare) să formeze prin fuziune celulară celule gigantice
multinucleate, denumite sincitiu[65-67].
Acest răspuns de fuziune celulară a depins de
scindarea dependentă de protează a proteinei spike în S1 și S2 și de scindarea
ulterioară a subunității S2 la locul S2′ [66] [4]. În final, aceste
sinciții suferă moarte celulară prin piroptoză, sporind răspunsul inflamator.
Mai mult, în cadrul sincițiilor, au fost detectate mai multe
micronuclee, iar aceste micronuclee au fost asociate cu γH2Ax (H2Ax cu Ser139
fosforilat), un marker extrem de precis și foarte timpuriu al deteriorării
ADN-ului [67]. Acești autori au scris: „Împreună, aceste rezultate sugerează
că micronucleii sincițiali sunt situsurile care cedează prin instabilitate genomică și leziuni ADN”.
[67] Creșterea detecției γH2AX prin exprimarea proteinei spike în celulele
epiteliale A549 este însoțită de o creștere a proteinelor supresoare tumorale
p16INK4A și a proteinelor oncogene p21 [63]. Faptul că proteina spike induce,
de asemenea, o suprareglare a LINE-1 ar trebui să ridice îngrijorare cu privire
la potențialul de revers-transcriere a ARNm-ului proteinei spike în contextul
formării de sinciții care invocă mecanismele de reparare a leziunilor ADN.
Așa-numita „cale de detectare a ADN-ului
cGAS-STING” este o cale biologică importantă care răspunde la fragmentele
de cromatină citoplasmatică (CCF) și activează un răspuns interferonic de tip
I. Sintetaza GMP-AMP ciclică (cGAS) este senzorul ADN care determină apoi
proteina din reticulul endoplasmatic, stimulatorul genelor interferonului
(STING), să declanșeze răspunsul interferonului. Calea cGAS-STING s-a
demonstrat a fi un actor-cheie în inducerea răspunsului interferonului de tip I
în celulele transfectate cu proteina spike [66] [4]. Autorii unui articol de
perspectivă privind calea cGAS-STING au scris: „În timp ce inflamația pe
termen scurt declanșată de calea CCF-cGAS-STING este necesară pentru eliminarea
imună a celulelor senescente, inflamația cronică mediată de SASP este
distructivă, având ca rezultat deteriorarea țesuturilor și chiar
tumorigeneza”. [68] O lucrare de analiză cu titlul provocator, „DNA Damage – How and Why We Age?” (Daunele ADN -ului – Cum și de ce
îmbătrânim?) a susținut că activarea excesivă a mecanismului de reparare a
ADN-ului din cauza deteriorării persistente a ADN-ului, poate fi cauza cauza
principală a îmbătrânirii accelerate și a bolilor asociate [69].
8. Mecanismele de reparare a ADN-ului: Un rol activ
pentru polimeraza Theta
În mod normal, atunci când o celulă este infectată cu un
virus, aceasta lansează imediat semnalizarea interferonului de tip I la
detectarea ARN-ului viral. Una dintre consecințele importante ale cascadei de
semnalizare este up-reglarea genei supresoare tumorale p53. P53 induce
oprirea ciclului celular la detectarea rupturilor ADN-ului dublu catenar,
protejând astfel celula de defecte genetice grave în timpul replicării și, prin
urmare, are un efect supresor tumoral. Sunt disponibile multiple
strategii de reparare a ADN-ului pentru a repara rupturile, astfel încât
replicarea să poată fi reluată. P53 oprește, de asemenea, replicarea
virală, încetinind astfel producția de copii multiple ale virusului limitând
răspândirea infecției [70]. Orice ruptură a catenei duble de ADN (DSB) deschide
oportunitatea unei translocații cromozomiale, în care cele două fragmente se
reatașează la cromozomi diferiți. Acest lucru poate avea ca rezultat atât lipsa
unor gene, cât și gene în plus, ceea ce poate perturba profund integritatea
cromozomială, provocând progresia către cancer. Astfel, este imperios necesar
să se repare ruptura înainte ca aceste potențial catastrofale modificări
genetice să aibă loc.
Editarea genelor este o tehnologie care oferă oamenilor de
știință capacitatea de a modifica ADNul unui organism, prin alterarea,
eliminarea sau inserarea unor secvențe genetice într-o anumită locație în
cadrul unui genom. Cea mai cunoscută tehnologie de editare genetică este CRISPR-Cas9
(clustered regularly interspaced short palindromic repeats – CRISPR-associated
protein 9). Această metodă este împrumutată din capacitate bacteriană de
combatere a bacteriofagilor. Secvențele CRISPR provenite din bacteriofagi, se
găsesc la jumătate din genomurile bacteriene secvențiate și în aproape 90% din
genomurile de archaea [71]. Cas9 utilizează secvențe CRISPR drept ghid pentru
recunoașterea și scindarea lanțurilor specifice de ADN care sunt complementare
cu șirul din secvența CRISPR.
Deși se consideră că CRISPR/Cas9 are o precizie ridicată asupra
locației din secvența de ADN pe care o modifică, acest lucru nu este
întotdeauna adevărat. Tehnologia constă în efectuarea unei rupturi a ADN-ului
dublu catenar și se bazează pe metodele celulare standard pentru repararea
rupturii. Celulele eucariote au dobândit mai multe mecanisme de reparare a
rupturilor de ADN, în funcție de stadiul ciclului celular. Așa-numita
recombinare omoloagă (HR) este foarte precisă, dar depinde de disponibilitatea
unui șablon de ADN ca ghid, care este disponibil doar în timpul ultimelor
stadii G2 și M. Joncțiunea neomologă a capătului (Nonhomologous end-joining NHEJ)
intră în joc în timpul etapelor anterioare ale ciclului celular (G0, G1 și S).
Procesul său de reparare este mai predispus la introducerea erorilor de
transcriere. Un al treilea mecanism de reparare, denumit microhomology-mediated
end joining (MMEJ), a fost recunoscut ca un mod alternativ de reparare abia în
ultimii ani [72]. Acesta implică mai întâi reconectarea a două regiuni
microhomologe din cele două șiruri rupte, iar apoi umplerea golurilor prin
sinteza ADN cu ajutorul unei ADN polimeraze.
O parte semnificativă din secvența originală este eliminată
prin acest proces și, prin urmare, este o soluție predispusă la erori.
Polimeraza theta (PolΘ) este o enzimă importantă de
reparare a ADN-ului, care implică repararea catenei duble de ADN, rupturi de ADN cu ajutorul MMEJ, cunoscută și
sub numele de „synthesis-dependent end joining” și „thetamediated end joining”. După cum s-a subliniat mai sus, metoda determină inserția de secvențe suplimentare
în locurile de îmbinare, precum și deleții – așa-numitele „indels”.
Aceasta nu este exprimată în majoritatea țesuturilor, tumorile fiind o excepție
notabilă. Este up-regulatătă în multe cancer, inclusiv în cancerul de stomac,
de plămâni și de colon, în cancerul de sân și cancerul ovarian, iar supraexpresia
sa reprezintă un prognostic pentru rezultate clinice slabe [73].
PolΘ este un factor cheie al evoluției genomului și al
mutagenezei mediate de CRISPR/Cas9 [74]. Am emis ipoteza că vaccinurile ARNm
care codifică proteina spike au creat o situație într-o celulă transfectată, în
special una aflată în stare proliferativă, care ar putea fi extrem de
susceptibilă la aberații cromozomiale severe. Deoarece tehnologia implică
modificări extinse ale ARNm viral original pentru a ascunde sursa virală,
aceasta realizează o intrare „invizibilă” în celulă fără a provoca un
răspuns normal al interferonului de tip I [75].
Celula declansează imediat o traducere eficientă a
ARNm pentru a produce cantități mari de proteină de spike. Proteina spike
provoacă leziuni grave ale ADN-ului, inclusiv rupturi ale catenei duble, așa
cum s-a descris anterior. Acest stres genetic lansează într-adevăr un răspuns
de interferon de tip I, dar acesta este întârziat, astfel încât daunele
semnificative au loc înainte ca p53 să fie suficient de crescută. Mai mult, afectarea
ADN-ului indusă de interferonul β este direct implicată în senescența celulară
și în inhibarea funcției celulelor stem, asociată cu îmbătrânirea accelerată [76].
O lucrare intitulată în mod corespunzător „Repair of G1
induced DNA double-strand breaks in S-G2/M by alternative NHEJ” (Repararea
rupturilor ADN-ului dublu-catenar induse de G1 în S-G2/M prin NHEJ alternativ)
a arătat, utilizând tehnologia CRISPR pentru a dezactiva p53, că rupturile de
ADN induse în timpul G1 ar putea fi reparate ulterior de polΘ, după ce ciclul
celular a avansat la faza S-G2/M [77]. Prin dezactivarea căii de reparare NHEJ,
mai puțin promiscuă, s-a permis ca ciclul celular să progreseze după ce ruptura
a avut loc, ceea ce a dus la generarea de multiple rearanjamente cromozomiale
aberante, promovând în același timp supraviețuirea globală a celulelor.
Capetele de ADN rupt în G1 generează translocații
cromozomiale cu o frecvență ridicată în timpul fazelor S-G2/M, indicând faptul
că aceste capete de ADN rupt și-au pierdut capacitatea de a se reconecta în
timpul progresiei ciclului celular. De reținut este că PolΘ nu este capabilă să
repare rupturile de ADN în timpul fazei G1 [77]. După cum au afirmat W. Feng și
colab., „dependența de Pol θ/TMEJ este asociată cu niveluri crescute de
DSBs (rupturi ale dublului lanț) asociate replicării, indiferent de deteriorarea
inițială”. [78] Acest lucru implică faptul că deteriorarea excesivă
a ADN-ului induce o suprareglare a polΘ. Acest lucru sugerează că celulele
canceroase și celulele imune proliferante transfectate cu proteina polΘ ar
suferi de o rată accelerată de mutații genetice, ceea ce ar duce la progresia
cancerului.
Un studiu publicat în 2021 a dezvăluit descoperirea
neașteptată că PolΘ este capabilă să transcrie invers ARN în ADN [7]. De fapt,
PolΘ prezintă o viteză și o fidelitate semnificativ mai mare de încorporare a
deoxiribonucleotidelor pe ARN decât pe ADN. Aceasta poate suferi o transformare
structurală remarcabilă pentru a menține interacțiuni productive pe șabloanele
ADN/ARN. Aceasta poate găzdui un hibrid complet ARN-ADN în situsul său activ și
poate transcrie eficient ribonucleotide șablon în ADN, promovând astfel
repararea ADN-ului pe bază de ARN. PolΘ pare a fi unică printre
polimerazele umane în ceea ce privește capacitatea sa de a transcrie invers
ARN, cu o eficiență echivalentă cu cea a retrovirusurilor. Prin urmare,
este posibil ca PolΘ să poată transcrie invers ARN-ul transpus de vaccin în ADN
și să îl integreze în genom în locurile de ruptură a ADN-ului.
Toate aceste considerații sunt rezumate în schema prezentată
în figura 2.
Figura 2: Schema evenimentelor care se presupune că se
desfășoară ca răspuns la absorbția celulară a secvențelor de ARNm din
vaccinurile cu ARNm ale SARS-CoV-2, în special în cazul celulelor cu un ciclu
celular activ. A se vedea textul pentru detalii.
9. Mecanisme de reparare a rupturilor ADN: Când ARN-ul
întâlnește ADN-ul.
Cu mult timp în urmă s-a postulat că, în afară de
retrovirusuri care au capacitatea de a se insera în ADN-ul uman prin
transcriere inversă, materialul genetic al tuturor celorlalte virusuri ARN nu
pot fi inserate în ADN în nicio circumstanță [1]. Cu toate acestea,
experimental, s-a dovedit de mult timp că nu este așa. În 2009, Geuking a
demonstrat o recombinare genetică, neexplicată altfel , ce are loc între virusul ARN al choriomeningitei
limfocitice și retrotranpozomul intracisternal endogen de tip A (IAP) și
aceasta a determinat revers-transcrierea ARN-ului viral exogen [11].
Acest ARN exogen a fost în cele din urmă inserat prin
intermediul ADN-ului său complementar în ADN-ul receptor, împreună cu elementul
IAP. De la această descoperire importantă, după cum au declarat autorii,
a devenit justificată investigarea adecvată a oricărei potențiale interacțiuni
cu elemente retrovirale, înainte ca virusurile ARN să poată fi utilizate în mod
terapeutic pentru inserția de material genetic nou.
Retroelementele sunt rămășițe active ale tranziției din lumea ARN în lumea ADN
care a avut loc cu milioane de ani în urmă pe Pământ. Interacțiunea activă a
tuturor elementelor genetice virale ARN cu ADN-ul eucariot este în prezent un
fenomen ușor de realizat, care susține biodiversitatea umană [17].
La potențialul problematic al integrării ARNm viral sau al
vaccinului în ADN-ul celulei gazdă se adaugă impactul potențial distructiv al
proteinei spike în sine asupra ADN-ului. Rupturile de ADN bicatenar sunt
un tip grav de deteriorare a ADN-ului și prezintă cel mai mare risc de inițiere
a unei transformări maligne în progeniturile celulelor afectate. BRCA1/2 și p53
orchestrează procese de reparare a ADN-ului extrem de complexe, orientate în
mod specific spre repararea rupturilor de ADN dsD [79].
Potențialul proteinei spike dea determina rupturi ale ADN-ului
dublu catenar pare convingător, având în vedere dovezile de formare de
micronuclee și sinciții în fibroblastele expuse [65-67]. Funcționarea
defectuoasă a punctelor de control G1 și G2/M este cuplată cu inhibarea
subnucleară a formării de focare BRCA1 și 53BP1 [9,80]. Un studiu in vitro a
arătat că subunitatea S2 a proteinei spike interacționează atât cu BRCA, cât și
cu p53, sugerând că ar putea interfera cu funcția lor anticancerigenă [81].
Celulele afectate de proteina spike a SARS-CoV-2 își continuă diviziunea
mitotică în ciuda rupturilor nereparate ale ADN-ului din cromozomi [82]. Acest
lucru favorizează evenimentele de recombinare, deoarece celulele sunt supuse
continuu transcripției și replicării, iar formarea buclelor R
cotranscripționale, dacă nu este regula, este iminentă [8].
Literatura de specialitate a descris efectele secundare ale proteinei
spike și ca urmare a vaccinării:
1. Afectarea endoteliului vascular [93]
2. Tromboză vasculară pulmonară și sistemică [94,95]
3. Tromboza venoasă și cerebrală [96]
4. Trombocitopenie, tromboembolism și hemoragie [97]
5. Defecte de agregare a trombocitelor [97]
6. Trombocitopenia imună severă refractară [98]
7. Miocardita [99]
8. Boala neurodegenerativă prionică [40,41]
9. Insuficiență multiorganică după vaccinarea împotriva
SARS-CoV-2 [100]
Figura 3. Modalități multiple prin care ARNm din vaccinurile
împotriva SARS-CoV-2 poate induce patologie și efecte secundare genetice în
celulele umane în diviziune și în organism. (A) Translația proteinei spike. Creșterea
translației ARNm-ului proteinei spike determină efecte adverse importante,
verificate în publicații, (B) Integrarea genomică; LINE-1, polimeraza theta și
retrotranspozonii autonomi cu revers transcriptază HERV pot, eventual, să
transcrie invers în cadrul celulelor vulnerabile ce se divid (celulele stem din
ganglionii limfatici) și să producă secvențe chimerice de fragmente de proteină
spike ale gazdei și virusului și noi virioni. Perturbarea genetică a
HERV-urilor altfel silențioase poate produce tulburări neurodegenerative și
cancer. (C) Reciclare; Degradarea continuă ARNm furnizează capete pentru
ARNm-ului endogen. (D). Complexele ARN-proteine; Fragmente de ARNm pentru proteina spike pot
forma complexe proteice cu nucleaze endogene interferând cu ADN-ul. LINE-1:
Elemente nucleare lungi intercalate 1; HERVS: retrovirusuri endogene umane; RT:
Transcripția inversă.
Efecte secundare descrise în literatura de specialitate
cauzate de proteina spike și de vaccinare:
1. Riscul de integrare în ADN-ul gazdei și de producere a de
transcriptelor chimerice [2,4,7].
2. Riscul producerii de virioni recombinanți [37,46]
3. Riscul producerii de celule stem canceroase și de
predispoziție către autoimunitate [54,62]
Literatura de specialitate publicată recent cu privire la
leziunile celulare și tisulare determinate de proteina spike a SARS-CoV-2
relevă un număr mare de leziuni cauzate de vaccinul COVID-19.
[40-41,93-100,102-106]. Multe dintre aceste leziuni pot fi așteptate dacă au
loc mecanismele descrise în această lucrare. Figura 2 rezumă constatările
noastre prin descrierea multiplelor moduri în care ARNm din vaccinurile
SARS-CoV-2 poate induce modificări patologice în celulele umane în diviziune.
Există dovezi solide că proteina spike în sine induce leziuni ale ADN-ului și
mecanismele ulterioare de reparare a ADN-ului. De asemenea, aceasta determină o
expresie crescută a LINE-1, care este capabilă să transforme ARNm în ADN.Transcripții chimerice pot apărea din procesele care au loc în nucleu. Complexele
ARN-proteină derivate din ARNm al vaccinului determină sechele imprevizibile. Aceste
procese combinate sugerează că expunerea la ARNm-ul care codifică proteina
Spike este potențial oncogenă, în special la cei care prezintă deja
polimorfisme ale p53 și/sau BRCA, precum
și la cei cu malignitate latentă sau manifestă.
10. Concluzie
Descoperirea recentă a integrării genomului SARS-CoV-2
[1,4,5,19] printr-un mecanism care implică LINE-1 sau polimeraza theta ridică
mari îngrijorări în ceea ce privește posibila încorporare nedorită și durabilă
a secvențelor proteice spike în genomul uman. Interferența ADN-ului
uman prin ARNm sintetic din vaccinuri este mai mult decât o simplă
posibilitate, deoarece transcrierea inversă a codului din ARNm al vaccinului
COVID-19 a fost demonstrată în liniile celulare hepatice canceroase umane.
Deoarece secvențele codificate de acestea sunt specifice pentru proteina spike
a SARS-CoV-2 și acestea pot fi, de asemenea, integrate în ADN-ul uman, patologia
rezultată ca urmare a vaccinării moleculare necesită o evaluare explicită prin
cercetări de genotoxicitate. În plus față de potențialul patogen al proteinei
spike codificate endogen (ADN), am demonstrat că activarea rețelelor enzimatice
celulare care realizează această integrare a ADN-ului au propriul lor potențial
patogen distinct și multifațetat. Se preconizează că aceste riscuri vor
fi mai ridicate în cazul unor populații vulnerabile specifice, și anume pentru persoanele
aflate în faza de dezvoltare (copii) și pacienții care suferă de cancere, boli
autoimune, boli cardiovasculare și neurologice și tulburări genetice.Recunoaștem că este speculativ să sugerăm că ARNm al vaccinului ar putea iniția
gama largă de evenimente patologice pe care le-am descris. Totuși, având
în vedere potențialul, documentat pe larg, al ARN-ului endogen (retroviral
uman) și exogen (viral) de a declanșa aceste evenimente, este nevoie urgentă de
investigații relevante, dat fiind numărul mare de persoane cărora li s-au
administrat unul sau mai multe produse ARNm care codifică proteina spike a
SARS-CoV-2.
Bibliografie
1. Pardi N, Hogan MJ, Porter FW, Weissman D. mRNA vaccines —
a new era in vaccinology. Nat Rev Drug Disc 2018; 17(4): 261-279. doi:
10.1038/nrd.2017.243.
2. Lehrer S, Rheinstein PH. SARS-CoV-2 orf1b gene sequence
in the NTNG1 gene on human chromosome 1. In Vivo 2020; 34(3 Suppl): 1629-1632.
doi: 10.21873/invivo.11953.
3. Ahmadpour D, Ahmadpoor P. How the COVID-19 overcomes the
battle? An approach to virus structure. Iran J Kidney Dis 2020; 14(3):
167-172.https://pubmed.ncbi.nlm.nih.gov/32361692/.
4. Lehrer S, Rheinstein PH. Human gene sequences in
SARS-CoV-2 and other viruses. In Vivo 2020; 34(3 Suppl): 1633-1636. doi:
10.21873/invivo.11954.
5. Zhang L, Richards AM, Barrasa MI, Hughes SH, Young RA,
Jaenisch R. Reversetranscribed SARS-CoV-2 RNA can integrate into the genome of
cultured human cells and can be expressed in patient-derived tissues. PNAS
2021; 118(21): e2105968118. doi: 10.1073/pnas.2105968118.
6. Weiner AM. SINEs and LINEs: The art of biting the hand
that feeds you. COCEBI 2002;14(3): 343-50. Doi: 10.1016/s0955-0674(02)00338-1.
7. Chandramouly G, Zhao J, McDevitt S, Rusanov T, Hoang T,
Borisonnik N, Treddinick T, Lopezcolorado FW, Kent T, Siddique LA, Mallon J,
Huhn J, Shoda Z, Kashkina E, Brambati A, Stark JM, Chen XS, Pomerantz RT. Polθ
reverse transcribes RNA and promotes RNA-templated DNA repair. Science Advances
2021; 7: eabf1771. doi: 10.1126/sciadv.abf1771.
8. Aguilera A, Gaillard H. Transcription and recombination:
When RNA meets DNA. Cold Spring Harb Perspect Biol 2014; 6: a016543. doi:
10.1101/cshperspect.a016543.
9. Liu J, Wang J, Xu J, Xia H, Wang Y, Zhang C, Chen W,
Zhang H, Liu Q, Zhu R, Shi Y, Shen Z, Xing Z, Gao W, Zhou L, Shao J, Shi J,
Yang X, Deng Y, Wu L, Lin Q, Zheng C, Zhu W, Wang C, Sun YE, Liu Z. Comprehensive
investigations revealed consistent pathophysiological alterations after
vaccination with COVID-19 vaccines. Cell Discovery 2021; 7: 99. doi:
10.1038/s41421-021-00329-3.
10. Katzourakis A, Gifford RJ. Endogenous viral elements in
animal genomes. PLoS Genet 2010; 6(11): e1001191. doi:
10.1371/journal.pgen.1001191.
11. Geuking MB, Weber J, Dewannieux M, Gorelik E, Heidmann
T, Hengartner H, Zinkernagel RM, Hangartner L. Recombination of retrotransposon
and exogenous RNA virus results in nonretroviral cDNA integration. Science
2009; 323(5912): 393-6. doi: 10.1126/science.1167375.
12. Franco-Muñoz C, Álvarez-Díaz DA, Laiton-Donato K,
Wiesner M, Escandón P, UsmeCiro JA, Franco-Sierra ND, Flórez-Sánchez AC,
Gómez-Rangel S, Rodríguez-Calderon LD, Barbosa-Ramirez J, Ospitia-Baez E,
Walteros DM, Ospina-Martinez ML, MercadoReyes M. Substitutions in spike and
nucleocapsid proteins of SARS-CoV-2 circulating in South America. Infect Genet
Evol 2020; 85: 104557. doi: 10.1016/j.meegid.2020.104557.
13. Grifoni A, Weiskopf D, Ramirez SK, Mateus J, Dan JM,
Moderbacher CR, Rawlings SA, Sutherland A, Premkumar L, Jadi RS, Marrama D, de
Silva AM, Frazier A, Carlin AF, Greenbaum JA, Peters B, Krammer F, Smith DM,
Crotty S, Sette A. Targets of T cell responses to SARS-CoV-2 coronavirus in
humans with COVID-19 disease and unexposed individuals. Cell 2020; 181(7):
1489-501. doi: 10.1016/j.cell.2020.05.015.
14. Pia L. SARS-CoV-2-reactive T cells in patients and
healthy donors. Nat Rev Immunol 2020; 20(6): 353. doi: 10.1038/s41586-020-2598-9.
15. Le Bert N, Tan AT, Kunasegaran K, Tham CYL, Hafezi M,
Chia A, Chng MHY, Lin M, Tan N, Linster M, Chia WN, Chen MI-C, Wang L-F, Ooi
EE, Kalimuddin S, Tambyah PA, Low JG-H, Tan Y-J, Bertoletti A.
SARS-CoV-2-specific T-cell immunity in cases of COVID-19 and SARS, and
uninfected controls. Nature 2020; 584(7821): 457-62. doi:
10.1038/s41586-020-2550-z.
16. Kyriakopoulos AM, Karafyllidou K, Li J, Zhao S.
Immuno-epidemiology and transmission characteristics of SARS-CoV-2: A
perspective investigation. J Clin Immunol Microbiol 2021; 2(2): 1-13. doi:
10.46889/JCIM.2021.2201.
17. Koonin EV, Dolja VV, Krupovic M. Origins and evolution
of viruses of eukaryotes: The ultimate modularity. Virology 2015; 479-480:
2-25. doi: 10.1016/j.virol.2015.02.039.
18. Pittoggi C, Beraldi R, Sciamanna I, Barberi L, Giordano
R, Magnano AR, Torosantucci L, Pescarmona E, Corrado Spadafora C. Generation of
biologically active retro-genes upon interaction of mouse spermatozoa with
exogenous dna. Molecular Reproduction and Development 2006; 73: 1239-1246. doi:
10.1002/mrd.20550.
19. Aldén M, Falla FO, Yang D, Barghouth M, Luan C,
Rasmussen M, De Marinis Y. Intracellular reverse transcription of Pfizer
BioNTech COVID-19 mRNA vaccine BNT162b2 in vitro in human liver cell line. Curr
Issues Mol Biol 2022; 44: 1115-1126. doi: 10.3390/cimb44030073.
20. Seneff S, Nigh G. Worse than the disease? Reviewing some
possible unintended consequences of the mRNA vaccines against COVID-19. IJVTPR
2021; 2(1): 38-79. https://ijvtpr.com/index.php/IJVTPR/article/view/23.
21. Bril F, Al Diffalha S, Dean M, Fettig DM. Autoimmune
hepatitis developing after coronavirus disease 2019 (COVID-19) vaccine:
Causality or casualty? J Hepatol 2021;75: 222–224. doi:
10.1016/j.jhep.2021.04.003.
22. Avci E, Abasiyanik F. Autoimmune hepatitis after
SARS-CoV-2 vaccine: New-onset or flare-up? J Autoimmun 2021; 125: 102745. doi:
10.1016/j.jaut.2021.102745.
23. McShane C, Kiat C, Rigby J, Crosbie Ó. The mRNA COVID-19
vaccine – A rare trigger of autoimmune hepatitis? J Hepatol 2021;75:
1239-1260. doi: 10.1016/j.jhep.2021.06.044.
24. Dai L, Taylor MS, O’Donnell KA, Boeke JD. Poly(A)
binding protein C1 is essential for efficient L1 retrotransposition and affects
L1 RNP formation. Mol Cell Biol 2012; 32:4323–4336. doi: 10.1128/MCB.06785-11.
25. Afonina E, Stauber R, Pavlakis GN. The human
poly(A)-binding protein 1 shuttles between the nucleus and the cytoplasm. J
Biol Chem 1998; 273: 13015-13021. doi:10.1074/jbc.273.21.13015.
26. Zhang X, Zhang R, Yu J. New understanding of the
relevant role of LINE-1 retrotransposition in human disease and immune
modulation. Front Cell Dev Biol 2020; 8; 657. doi: 10.3389/fcell.2020.00657.
27. Honda T. Links between human LINE-1 retrotransposons and
hepatitis virus-related hepatocellular carcinoma. Front Chem 2018; 4: 21. Doi:
10.3389/fchem.2016.00021.
28. Silva E, Marques S, Osório H, Carvalheira J, Thompson G.
Endogenous hepatitis C virus homolog fragments in European rabbit and hare genomes
replicate in cell culture. PLoS One 2012; 7(11): e49820. doi:
10.1371/journal.pone.0049820.
29. Beard MR, Abell G, Honda M, Carroll A, Gartland M,
Clarke B, Suzuki K, Lanford R, Sangar DV, Lemon, SM. An infectious molecular
clone of a Japanese genotype 1b hepatitis C virus. Hepatology 1999; 30(1):
316-324. doi: 10.1002/hep.510300137.
30. Zhang Y-J, Wu H-C, Yazici H, Yu M-W, Lee P-H, Santella
RM. Global hypomethylation in hepatocellular carcinoma and its relationship to
aflatoxin B1 exposure. World J Hepatol 2012; 4(5): 169-175. doi:
10.4254/wjh.v4.i5.169.
31. Zhuo C, Li Q, Wu Y, Li Y, Nie J, Li D, Peng J, Lian P,
Li B, Cai G, Li X, Cai S. LINE-1 hypomethylation in normal colon mucosa is
associated with poor survival in Chinese patients with sporadic colon cancer.
Oncotarget 2015; 6: 23820-23836. doi: 10.18632/oncotarget.4450.
32. Rastogi M, Pandey N, Shukla A, Singh SK. SARS
coronavirus 2: from genome to infectome. Respiratory Research 2020; 21: 318.
doi: 10.1186/s12931-020-01581-z.
33. Aoki-Suzuki M, Yamada K, Meerabux J, Iwayama-Shigeno Y,
Ohba H, Iwamoto K, Takao H, Toyota T, Suto Y, Nakatani N, Dean B, Nishimura S,
Seki K, Kato T, Itohara S, Nishikawa T, Yoshikawa Y. A family-based association
study and gene expression analyses of netrin-G1 and -G2 genes in schizophrenia.
Comparative Study Biol Psychiatry 2005; 57(4): 382-93. doi:
10.1016/j.biopsych.2004.11.022.
34. Eastwood SL, Harrison PJ. Decreased mRNA expression of
netrin-G1 and netrin-G2 in the temporal lobe in schizophrenia and bipolar
disorder. Neuropsychopharmacology 2008; 33: 933-945. doi:
10.1038/sj.npp.1301457.
35. Xiao R, Chen JY, Liang Z, Luo D, Chen G, Lu ZJ, Chen Y,
Zhou B, Li H, Du X, Yang Y, San M, Wei X, Liu W, Lécuyer E, Graveley BR, Yeo
GW, Burge CB, Zhang MQ, Zhou Y, Fu XD. Pervasive chromatin-RNA binding protein
interactions enable RNA-based regulation of transcription. Cell 2019; 178(1):
107-121.e18. doi: 10.1016/j.cell.2019.06.001.
36. Ahmad M, Shen W, Li W, Xue Y, Zou S, Xu D, Wang W.
Topoisomerase 3β is the major topoisomerase for mRNAs and linked to
neurodevelopment and mental dysfunction. Nucleic Acids Res 2017; 45(5):
2704-2713. doi: 10.1093/nar/gkw1293.
37. Poltronieri P, Sun B, Mallardo, M. RNA viruses: RNA
roles in pathogenesis, coreplication and viral load. Curr Genomics 2015; 5:
327-35. doi: 10.2174/1389202916666150707160613.
38. Küry P, Nath A, Créange A, Dolei A, Marche P, Gold J,
Giovannoni G, Hartung H-P, Perron H. Human endogenous retroviruses in
neurological diseases. Trends Mol Med 2018; 24(4): 379-394. doi: 10.1016/j.molmed.2018.02.007.
39. Erro R, Buonomo AR, Barone P. Pellecchia, M.T. Severe
dyskinesia after administration of SARS-CoV2 mRNA vaccine in Parkinson’s
disease. Movement Disorders 2021; 36: 10, 2219. doi: 10.1002/mds.28772.
40. Classen JB. COVID-19 vaccine associated Parkinson’s
disease, A prion disease signal in the UK Yellow Card adverse event database. J
Med – Clin Res & Rev 2021; 5(7):
1-6.https://scivisionpub.com/abstract-display.php?id=1746.
41. Classen JB. COVID-19 RNA based vaccines and the risk of
prion disease. Microbiol Infect Dis 2021; 5(1): 1-3.
42. Patterson BK, Francisco EB, Yogendra R, Long E, Pise A,
Rodrigues H, Hall E, Herrara M, Parikh P, Guevara-Coto J, Triche TJ, Scott P,
Hekmati S, Maglinte D, Chang X, Chang X, Mora-Rodríguez RA, Mora J. Persistence
of SARS CoV-2 S1 protein in CD16+ monocytes in post-acute sequelae of COVID-19
(PASC) up to 15 months post-infection. Front Immunol 2022; 12: 746021. doi:
10.3389/fimmu.2021.746021.
43. Röltgen K, Nielsen SCA, Silva O, Younes SF, Zaslavsky M,
Costales C, Yang F, Wirz OF, Solis D, Hoh RA, Wang A, Arunachalam PS, Colburg
D, Zhao S, Haraguchi E, Lee AS, Shah MM, Manohar M, Chang I, Gao F,
Mallajosyula V, Li C, Liu J, Shoura MJ, Sindher SB, Parsons E, Dashdorj NJ,
Dashdorj ND, Monroe R, Serrano GE, Beach TG, Chinthrajah RS, Charville GW,
Wilbur JL, Wohlstadter JN, Davis MM, Pulendran B, Troxell ML, Sigal GB,
Natkunam Y, Pinsky BA, Nadeau KC, Boyd SD. Immune imprinting, breadth of
variant recognition, and germinal center response in human SARS-CoV-2 infection
and vaccination. Cell 2022; 185: 1-16. doi: 10.1016/j.cell.2022.01.018.
44. Chetverin AB. Replicable and recombinogenic RNAs. FEBS
Lett 2004; 567(1): 35-41. doi: 10.1016/j.febslet.2004.03.066.
45. Jäschke A, Helm M. RNA sex. Chem Biol 2003; 10(12):
1148-50. doi: 10.1016/j.chembiol.2003.12.003.
46. Hughes S.H. Reverse transcription of retroviruses and
LTR retrotransposons. Microbiol Spectr 2015; 3(2): MDNA3-2014. doi:
10.1128/microbiolspec.MDNA3-0027-2014.
47. Vitullo P, Sciamanna I, Baiocchi M, Sinibaldi-Vallebona
P, Spadafora C. LINE-1 retrotransposon copies are amplified during murine early
embryo development. Mol Reprod Dev 2012; 79(2): 118-27. doi: 10.1002/mrd.22003.
48. Katoh I. Impacts of endogenous retroviruses on
tumorigenesis, immunity, stem cells, and research safety. Front Oncol 2014; 4:
66. doi: 10.3389/fonc.2014.00066.
49. Hohn O, Hanke K, Bannert N. HERV-K(HML-2), the best preserved
family of HERVs: Endogenization, expression, and implications in health and
disease. Front Oncol 2013;3: 246. doi: 10.3389/fonc.2013.00246.
50. Golombek S, Pilz M, Steinle H, Kochba E, Levin Y, Lunter
D, Schlensak C, Wendel HP, Avci-Adali M. Intradermal delivery of synthetic mRNA
using hollow microneedles for efficient and rapid production of exogenous
proteins in skin. Mol Ther Nucleic Acids 2018; 11: 382-392. doi:
10.1016/j.omtn.2018.03.005.
51. DNA and RNA Molecular Weights and Conversions. ThermoFisher
Scientific.
https://www.thermofisher.com/gr/en/home/references/ambion-tech-support/rnatools-and-calculators/dna-and-rna-molecular-weights-and-conversions.html
52. Ols S, Loré K. Imaging the early fate of mRNA vaccines.
Nat Biomed Eng 2019; 3(5):331-332. doi: 10.1038/s41551-019-0399-y.
53. Capece T, Kim M. The role of lymphatic niches in T cell
differentiation. Mol Cells 2016;39(7): 515-523. doi:
10.14348/molcells.2016.0089.
54. Jiang Q, Crews LA, Holm F, Jamieson CHM. RNA
editing-dependent epitranscriptome diversity in cancer stem cells. Nat Rev
Cancer 2017; 17(6): 381-392. doi: 10.1038/nrc.2017.23.
55. Patra T, Meyer K, Geerling L, Isbell TS, Hoft DF, Brien
J, Pinto AK, Ray RB, Ray R. SARS-CoV-2 spike protein promotes IL-6
trans-signaling by activation of angiotensin II receptor signaling in
epithelial cells. PLoS Pathog 2020; 16(12): e1009128. doi:
10.1371/journal.ppat.1009128.
56. Khan S, Shafiei MS, Longoria C, Schoggins JW, Savani RC,
Zaki H. SARS-CoV-2 spike protein induces inflammation via TLR2-dependent
activation of the NF-κB pathway. eLife 2021; 10: e68563. doi:
10.7554/eLife.68563.
57. Wang W, Ye L, Ye L, Li B, Gao B, Zeng Y, Kong L, Fang X,
Zheng H, Wu Z, She Y. Up-regulation of IL-6 and TNF-alpha induced by
SARS-coronavirus spike protein in murine macrophages via NF-kappaB pathway.
Virus Res 2007, 128(1-2), 1-8. doi: 10.1016/j.virusres.2007.02.007.
58. Johnson DE, O’Keefe RA, Grandis, JR. Targeting the
IL-6/JAK/STAT3 signalling axis in cancer. Nat Rev Clin Oncol. 2018; 15(4):
234–248. doi: 10.1038/nrclinonc.2018.8.35
59. Jiang Q, Isquith J, Ladel L, Mark A, Holm F, Mason C, He
Y, Mondala P, Oliver I, Pham J, Ma W, Reynoso E, Ali S, Morris IJ, Diep R,
Nasamran C, Xu G, Sasik R, Rosenthal SB, Birmingham A, Coso S, Pineda G, Crews
L, Donohoe ME, Venter JC, Whisenant T, Mesa RA, Alexandrov LB, Fisch KM,
Jamieson C. Inflammation-driven deaminase deregulation fuels human pre-leukemia
stem cell evolution. Cell Reports 2021; 34: 108670. doi:
10.1016/j.celrep.2020.108670.
60. Vlachogiannis NI, Verrou K-M, Stellos K, Sfikakis PP,
Paraskevisd D. The role of A-toI RNA editing infections by RNA viruses:
Possible implications for SARS-CoV-2 infection. Clinical Immunology 2021; 226:
108699. doi: 10.1016/j.clim.2021.108699.
61. Dominissini D, Moshitch-Moshkovitz S, Amariglio N,
Rechavi G. Adenosine-toinosine RNA editing meets cancer. Carcinogenesis 2011;
32(11): 1569-1577. doi: 10.1093/carcin/bgr124.
62. Clément F, Grockowiak E, Zylbersztejn F, Fossard G,
Gobert S, Maguer-Satta V. Stem cell manipulation, gene therapy and the risk of
cancer stem cell emergence. Stem Cell Investig 2017; 4: 67. doi:
10.21037/sci.2017.07.03
63. Meyer K, Patra T, Vijayamahantesh, Ray R. SARS-CoV-2
spike protein induces paracrine senescence and leukocyte adhesion in
endothelial cells. J Virol 2021; 95(17): e0079421. doi: 10.1128/JVI.00794-21
64. Li F, Li J, Wang PH, Yang N, Huang J, Ou J, Xu T, Zhao X, Liu T, Huang X,
Wang Q, Li M, Yang L, Lin Y, Cai Y, Chen H, Zhang Q. SARS-CoV-2 spike promotes
inflammation and apoptosis through autophagy by ROS-suppressed PI3K/AKT/mTOR
signaling. Biochim Biophys Acta Mol Basis Dis 2021; 1867(12): 166260. doi:
10.1016/j.bbadis.2021.166260.
65. Ma H, Zhu Z, Lin H, Wang S, Zhang P, Li Y, Li L, Wang J,
Zhao Y, Han J. Pyroptosis of syncytia formed by fusion of SARS-CoV-2 spike and
ACE2-expressing cells. Cell Discov 2021; 7: 73. doi: 0.1038/s41421-021-00310-0.
66. Liu X, Wei L, Xu F, Zhao F, Huang Y, Fan Z, Mei S, Hu Y,
Zhai L, Guo J, Zheng A, Cen S, Liang C, Guo F. SARS-CoV-2 spike protein-induced
cell fusion activates the cGASSTING pathway and the interferon response. Sci
Signal 2022; 15(729): eabg8744. doi: 10.1126/scisignal.abg8744.
67. Ren H, Ma C, Peng H, Zhang B, Zhou L, Su Y, Gao X, Huang
H. Micronucleus production, activation of DNA damage response and cGAS-STING
signaling in syncytia induced by SARS-CoV-2 infection. Biol Direct 2021; 16:
20. doi: 10.1186/s13062-021-00305-7.
68. Chen K, Liu J, Cao,X. cGAS-STING pathway in
senescence-related inflammation. Natl Sci Rev 2018; 5(3): 308-310. doi: 10.1093/nsr/nwx146.
69. Yousefzadeh M, Henpita C, Vyas R, Soto-Palma C, Robbins
P, Niedernhofer L. DNA damage-how and why we age? Elife 2021; 10: e62852. doi:
10.7554/eLife.62852.
70. Muñoz-Fontela C, Macip S, Martínez-Sobrido L, Brown L,
Ashour J, García-Sastre A, Lee SW, Aaronson SA. Transcriptional role of p53 in
interferon-mediated antiviral immunity. J Exp Med 2008; 205(8): 929-1938.
doi:10.1084/jem.20080383.
71. Hille F, Richter H, Wong SP, Bratovič M, Ressel S,
Charpentier E. The biology of CRISPR-Cas: backward and forward. Cell 2018; 172
(6): 12391259. doi: 10.1016/j.cell.2017.11.032.
72. Wang H, Xu X. Microhomology-mediated end joining: new
players join the team. Cell Biosci 2017; 7: 6. doi: https://doi.org/10.1186/s13578-017-0136-8.
73. Wood RD, Doublié S. DNA polymerase (POLQ), double-strand
break repair, and cancer DNA Repair (Amst). 2016; 44: 22-32. doi:
10.1016/j.dnarep.2016.05.003.
74. Schendel R, Roerink SF, Portegijs V, van den Heuvel S,
Tijsterman M. Polymerase Θ is a key driver of genome evolution and of
CRISPR/Cas9-mediated mutagenesis. Nature Communications 2015; 6: 7394. doi:
10.1038/ncomms8394.
75. Seneff S, Nigh G, Kyriakopoulos, AM, McCullough PA. Innate
immune suppression by SARS-CoV-2 mRNA vaccinations: The role of G-quadruplexes,
exosomes, and MicroRNAs. Food and Chemical Toxicology 2022; 164: 113008. doi:
https://doi.org/10.1016/j.fct.2022.113008.
76. Yu Q, Katlinskaya YV, Carbone CJ, Zhao B, Katlinski KV,
Zheng H, Guha M, Li N, Chen Q, Yang T, Lengner CJ, Greenberg RA, Johnson FB,
Fuchs SY. DNA damageinduced type I interferon promotes senescence and inhibits
stem cell function Cell Rep 2015; 11(5): 785-797. doi:
10.1016/j.celrep.2015.03.069.
77. Yu W, Lescale C, Babin L, Bedora-Faure M, Lenden-Hasse
H, Baron L, Demangel C, Yelamos J, Brunet E, Deriano L. Repair of G1 induced
DNA double-strand breaks in SG2/M by alternative NHEJ. Nat Commun 2020; 11:
5239. doi: https://doi.org/10.1038/s41467-020-19060-w.
78. Simpson DA, Carvajal-Garcia J, Price BA, Kumar RJ, Mose
LE, Wood RD, Rashid N, Purvis JE, Parker JS, Ramsden DA. Genetic determinants
of cellular addiction to DNA polymerase theta. Nature Communications 2019; 10:
4286. doi: 10.1038/s41467-019-12234-1.
79. Khanna K, Jackson S. DNA double-strand breaks:
signaling, repair and the cancer connection. Nat Genet 2001; 27: 247-254. doi:
https://doi.org/10.1038/85798].
80. Daley JM, Sung P. 53BP1, BRCA1, and the choice between
recombination and end joining at DNA double-strand breaks. Mol Cell Biol 2014;
34(8): 1380-1388. doi:10.1128/MCB.01639-13.
81. Singh N, Singh AB. S2 Subunit of SARS-nCoV-2 interacts
with tumor suppressor protein p53 and BRCA: an in silico study. Translational
Oncology 2020; 13: 100814. doi: 10.1016/j.tranon.2020.100814.
82. Brown EJ, Baltimore D. Essential and dispensable roles
of ATR in cell cycle arrest and genome maintenance. Genes Dev 2003; 17(5):
615-28. doi: 10.1101/gad.1067403.
83. Huertas P. DNA resection in eukaryotes: deciding how to
fix the break. Nat Struct Mol Biol 2010; 17(1): 11-16. doi:10.1038/nsmb.1710.
84. Nakamura K, Saredi G, Becker JR, Foster BM, Nguyen NV,
Beyer TE, Cesa LC, Faull PA, Lukauskas S, Frimurer T, Chapman JR, Bartke T,
Groth A. H4K20me0 recognition by BRCA1-BARD1 directs homologous recombination
to sister chromatids. Nat Cell Biol 2019; 21(3): 311-318. doi:
10.1038/s41556-019-0282-9.
85. Harris JL, Khanna KK. BRCA1 A-complex fine tunes repair
functions of BRCA1. Aging (Albany NY) 2011; 3(5): 461-463.
doi:10.18632/aging.100334.
86. Dever SM, Golding SE, Rosenberg E, Adams BR, Idowu MO,
Quillin JM, Valerie N, Xu B, Povirk LF, Valerie K. Mutations in the BRCT
binding site of BRCA1 result in hyper-recombination. Aging (Albany NY) 2011;
3(5): 515-32. doi: 10.18632/aging.100325.
87. Zhang R, Xiao K, Gu Y, Liu H, Sun X. Whole genome
identification of potential Gquadruplexes and analysis of the G-quadruplex
binding domain for SARS-CoV-2. Front Genet 2020; 11: 587829. doi:
10.3389/fgene.2020.587829.
88. McKernan K, Kyriakopoulos AM, McCullough PA. Differences
in vaccine and SARSCoV-2 replication derived mRNA: Implications for cell
biology and future disease. OSF Preprint. Nov 24, 2021. doi:
10.31219/osf.io/bcsa6 https://osf.io/bcsa6/
89. Allison DF, Wang GG. R-loops: formation, function, and
relevance to cell stress. Cell Stress 2019; 3(2): 38-46. doi:
10.15698/cst2019.02.175.
90. Seinsoth S, Uhlmann-Schiffler H,Stahl H. Bidirectional
DNA unwinding by a ternary complex of T antigen, nucleolin and topoisomerase I.
EMBO Rep 2003; 4(3): 263-268. doi:10.1038/sj.embor.embor770.
91. Zhang J. Host RNA polymerase II makes minimal
contributions to retroviral frameshift mutations. J Gen Virol 2004; 85(Pt 8):
2389-2395. doi: 10.1099/vir.0.80081-0.
92. Mohammed SI, Torres-Luquis O, Walls E, Lloyd F.
Lymph-circulating tumor cells show distinct properties to blood-circulating
tumor cells and are efficient metastatic precursors. Mol Oncol 2019; 13(6):
1400-1418. doi: 10.1002/1878-0261.12494.
93. Lei Y, Zhang J, Schiavon CR, He M, Chen L, Shen H, Zhang
Y, Yin Q, Cho Y, Andrade L, Shadel GS, Hepokoski M, Lei T, Wang H, Zhang J,
Yuan J X-J, Malhotra A, Manor U, Wang S, Yuan Z-Y, Shyy J Y-J. SARS-CoV-2 spike
protein impairs endothelial function via downregulation of ACE2. Circ Res 2021;
128(9): 1323-1326. doi: 10.1161/CIRCRESAHA.121.318902.
94. Zhang J, Tecson KM, McCullough PA. Role of endothelial
cell receptors in the contextof SARS-CoV-2 infection (COVID-19). Proc (Bayl
Univ Med Cent) 2021; 34(2): 262-268. doi: 10.1080/08998280.2021.1874231.
95. Zhang J, Tecson KM, McCullough PA. Endothelial
dysfunction contributes to COVID19-associated vascular inflammation and
coagulopathy. Rev Cardiovasc Med 2020;21(3): 315-319. doi:
10.31083/j.rcm.2020.03.126.
96. See I, Su JR, Lale A, Woo EJ, Guh AY, Shimabukuro TT,
Streiff MB, Rao AK, Wheeler AP, Beavers SF, Durbin AP, Edwards K, Miller E,
Harrington TA, Mba-Jonas A, Nair N, Nguyen DT, Talaat KR, Urrutia VC, Walker
SC, Creech CB, Clark TA, DeStefano F, Broder KR. US case reports of cerebral
venous sinus thrombosis with thrombocytopenia after Ad26.COV2.S vaccination.
JAMA 2021; 325(24): 2448-2456.
doi: 10.1001/jama.2021.7517.
97. Simpson CR, Shi T, Vasileiou E, Katikireddi SV, Kerr S,
Moore E, McCowan C, Agrawal U, Shah SA, Ritchie LD, Murray J, Pan J, Bradley
DT, Stock SJ, Wood R, Chuter A, Beggs J, Stagg HR, Joy M, Tsang RSM, de
Lusignan S, Hobbs R, Lyons RA, Torabi F, Bedston S, O’Leary M, Akbari A,
McMenamin J, Robertson C, Sheikh A. Firstdose ChAdOx1 and BNT162b2 COVID-19
vaccines and hrombocytopenic,
thromboembolic and hemorrhagic events in Scotland. Nat Med
2021; 27: 1290-1297. doi: 10.1038/s41591-021-01408-4.
98. Helms JM, Ansteatt KT, Roberts JC, Kamatam S, Foong KS,
Labayog J-MS, Tarantino MD. Severe, refractory immune thrombocytopenia
occurring after SARS-CoV-2 vaccine. J Blood Med 2021; 12: 221-224. doi:
10.2147/JBM.S307047.
99. Montgomery J, Ryan M, Engler R, Hoffman D, McClenathan
B, Collins L, Loran D, Hrncir D, Herring K, Platzer M, Adams N, Sanou A, Cooper
LT, Jr. Myocarditis following immunization with mRNA COVID-19 vaccines in
members of the US military. JAMA Cardiol 2021; 6(10): 1202-1206. doi:
10.1001/jamacardio.2021.2833.
100. Hansen T, Titze U, Kulamadayil-Heidenreich, NSA,
Glombitz S, Tebbe JJ, Röcken C, Schulz B, Weise M, Wilkens L. First case of
postmortem study in a patient vaccinated against SARS-CoV-2. Int J Infect Dis
2021; 107: 172–175. doi: 10.1016/j.ijid.2021.04.053.
101. Liu Y, Soh WT, Tada A, Arakawa A, Matsuoka S, Nakayama
EE, Li S, Ono C, Torii S, Kishida K, Jin H, Nakai W, Arase N, Nakagawa A,
Shindo Y, Kohyama M, Nakagami H, Tomii K, Ohmura K, Ohshima S, Okada M,
Matsuura Y, Standley DM, Shioda Y, Arase H. An infectivity-enhancing site on
the SARS-CoV-2 spike protein targeted by antibodies. Cell 2021; 184(13):
3452-3466. doi: 0.1016/j.cell.2021.05.032.
102. Holtkamp S, Kreiter S, Selmi A, Simon P, Koslowski M,
Huber C, Türeci O, Sahin U. Modification of antigen-encoding RNA increases
stability, translational efficacy, and T-cell stimulatory capacity of dendritic
cells. Blood 2006; 108(13): 4009-17. doi: 10.1182/blood-2006-04-015024.
103. Strenkowska M, Grzela R, Majewski M, Wnek K, Kowalska
J, Lukaszewicz M, Zuberek J, Darzynkiewicz E, Kuhn AN, Sahin U, Jemielity J.
Cap analogs modified with 1,2-dithiodiphosphate moiety protect mRNA from
decapping and enhance its translational potential. Nucleic Acids Res 2016;
44(20): 9578-9590. doi: 10.1093/nar/gkw896.
104. Ramanathan A, Robb GB, Chan S-H. mRNA capping:
biological functions and applications, Nucleic Acids Research 2016; 44(16):
7511-26. doi: 10.1093/nar/gkw551.
105. Balagopal V, Parker R. Polysomes, P bodies and stress
granules: states and fates of eukaryotic mRNAs. Curr Opin Cell Biol 2009; (3):
403-8. doi: 10.1016/j.ceb.2009.03.005.
106. Kyriakopoulos AM, McCullough PA. Synthetic mRNAs; Their
analogue caps and contribution to disease. Diseases 2021; 9(3): 57. doi:
10.3390/diseases9030057